KLEINER RÜCKBLICK
Der Vorposten gegen Krebs: Universelle Krebsmarker
Chengchen Qian1, Xiaolong Zou2, Wei Li1,3, Yinshan Li4, Wenqiang Yu5
1Shanghai Epiprobe Biotechnology Co., Ltd, Shanghai 200233, China;2 Abteilung für Allgemeine Chirurgie, das erste angegliederte Krankenhaus der Harbin Medical University, Harbin 150001, China;3Shandong Epiprobe Medical Laboratory Co., Ltd, Heze 274108, China;4 Volkskrankenhaus der Autonomen Region Ningxia Hui, Medizinische Universität Ningxia, Yinchuan 750002, China;5Shanghai Public Health Clinical Center & Abteilung für Allgemeine Chirurgie, Huashan Hospital & Cancer Metastasis Institute & Laboratory of RNA Epigenetics, Institutes of Biomedical Sciences, Shanghai Medical College, Fudan University, Shanghai 200032, China
ABSTRAKT
Krebs ist weltweit die häufigste Todesursache.Die Früherkennung von Krebs kann die Sterblichkeit aller Krebsarten senken;Für die meisten Krebsarten fehlen jedoch wirksame Biomarker zur Früherkennung.Die DNA-Methylierung war schon immer ein wichtiges Ziel, da die DNA-Methylierung normalerweise vor anderen nachweisbaren genetischen Veränderungen erfolgt.Bei der Untersuchung der gemeinsamen Merkmale von Krebs mithilfe eines neuartigen Leitfadens, der die Sequenzierung für die DNA-Methylierung positioniert, hat sich eine Reihe von universellen Nur-Krebs-Markern (UCOMs) als starke Kandidaten für eine wirksame und genaue Früherkennung von Krebs herausgestellt.Während der klinische Wert aktueller Krebsbiomarker durch geringe Sensitivität und/oder geringe Spezifität gemindert wird, gewährleisten die einzigartigen Eigenschaften von UCOMs klinisch bedeutsame Ergebnisse.Die Validierung des klinischen Potenzials von UCOMs bei Lungen-, Gebärmutterhals-, Endometrium- und Urothelkrebs unterstützt zusätzlich die Anwendung von UCOMs bei mehreren Krebsarten und verschiedenen klinischen Szenarien.Tatsächlich werden die Anwendungen von UCOMs derzeit aktiv untersucht und in den Bereichen Früherkennung von Krebs, Hilfsdiagnose, Behandlungswirksamkeit und Rezidivüberwachung weiter evaluiert.Die molekularen Mechanismen, mit denen UCOMs Krebs erkennen, sind die nächsten wichtigen Themen, die untersucht werden müssen.Auch die Anwendung von UCOMs in realen Szenarien erfordert Implementierung und Verfeinerung.
SCHLÜSSELWÖRTER
Krebserkennung;Krebsvorsorge;DNA-Methylierung;Krebsepigenetik;Krebsbiomarker
Warum wir dringend Neues brauchen Biomarker?
Nach über einem Jahrhundert Kampf gegen Krebs ist Krebs immer noch die tödlichste biologische Bedrohung für die Menschheit.Krebs bleibt ein globales Gesundheitsproblem mit schätzungsweise 19,3 Millionen neuen Fällen und fast 10 Millionen Todesfällen im Jahr 20201. Im Jahr 2020 wurden in China schätzungsweise 4,6 Millionen neue Krebsfälle diagnostiziert, was laut GLOBOCAN1 23,7 % aller neuen Krebsfälle weltweit ausmacht.Darüber hinaus wurden im Jahr 2020 in China etwa 3 Millionen Todesfälle auf Krebs zurückgeführt, was 30 % der weltweiten krebsbedingten Todesfälle ausmacht1.Diese Statistiken zeigen, dass China bei der Inzidenz- und Sterblichkeitsrate von Krebs an erster Stelle steht.Darüber hinaus beträgt die 5-Jahres-Überlebensrate bei Krebs 40,5 %, was 1,5-mal niedriger ist als die 5-Jahres-Überlebensrate in den Vereinigten Staaten2,3.Die vergleichsweise niedrigeren Überlebens- und höheren Sterblichkeitsraten in China als in Ländern mit höheren menschlichen Entwicklungsindizes legen nahe, dass ein effizientes und kostengünstiges System zur Krebsprävention und -überwachung dringend erforderlich ist.Die Früherkennung von Krebs ist eines der wichtigsten Elemente im Gesundheitssystem.Die Früherkennung von Krebs kann bei fast allen Krebsarten die Prognose und das Überleben in einem frühen Stadium verbessern4.Erfolgreiche Screening-Strategien haben zu einem deutlichen Rückgang der Inzidenz- und Mortalitätsraten von Gebärmutterhals-, Brust-, Darm- und Prostatakrebs geführt.
Eine Krebsfrüherkennung zu erreichen, ist jedoch keine leichte Aufgabe.Die Erforschung der Biologie und Prognose von Krebs im Frühstadium, die Identifizierung und Validierung zuverlässiger Früherkennungs-Biomarker sowie die Entwicklung zugänglicher und genauer Früherkennungstechnologien waren schon immer die größten Hindernisse in diesem Prozess4.Durch die präzise Erkennung von Krebs können gutartige von bösartigen Läsionen unterschieden werden, was dazu beiträgt, unnötige Eingriffe zu vermeiden und die weitere Behandlung der Krankheit zu erleichtern.Aktuelle Früherkennungsstrategien umfassen endoskopbasierte Biopsien, medizinische Bildgebung, Zytologie, Immunoassays und Biomarkertests5-7.Da endoskopbasierte Biopsien aufwendig und kostspielig sind, stellen sie als große medizinische Prozedur, die auf professionelles Personal angewiesen ist, eine große Belastung dar.Wie die Zytologie sind auch beide Screening-Methoden von medizinischem Fachpersonal abhängig und basieren auf einer persönlichen Beurteilung, wobei die Leistung alles andere als ideal ist8.Im Gegensatz dazu sind Immunoassays angesichts der hohen Falsch-Positiv-Raten äußerst ungenau.Medizinische Bildgebung als Screening-Taktik erfordert teure Ausrüstung und spezialisierte Techniker.Daher ist die medizinische Bildgebung aufgrund der geringen Zugänglichkeit äußerst eingeschränkt.Aus all diesen Gründen scheinen Biomarker eine bessere Option für die Früherkennung von Krebs zu sein.
Korrespondenz mit: Yinshan Li und Wenqiang Yu
Email: liyinshan@nxrmyy.com and wenqiangyu@fudan.edu.cn
ORCID-ID: https://orcid.org/0009-0005-3340-6802 und
https://orcid.org/0000-0001-9920-1133
Eingegangen am 22. August 2023;angenommen am 12. Oktober 2023;
online veröffentlicht am 28. November 2023.
Verfügbar unter www.cancerbiomed.org
©2023 Krebsbiologie und Medizin.Creative Commons
Namensnennung – nicht kommerziell 4.0 Internationale Lizenz
Biomarker werden derzeit in Proteine, DNA-Mutationsmarker, epigenetische Marker, Chromosomenanomalien, direkt aus Tumoren stammende RNA-Marker oder indirekt aus Körperflüssigkeiten gewonnene Tumorfragmente eingeteilt.Proteinmarker sind die am häufigsten verwendeten Biomarker bei der Krebsvorsorge und -diagnose.Protein-Biomarker sind als Screening-Biomarker durch die Tendenz eingeschränkt, von gutartigen Läsionen betroffen zu sein, was zu Überdiagnose und Überbehandlung führt, wie für α-Fetoprotein und Prostata-spezifisches Antigen (PSA) berichtet wurde9,10.Zu den RNA-Markern gehören genetische Expressionsmuster und andere nichtkodierende RNA-Marker. Eine Kombination von genetischen Expressions-RNA-Markern kann mithilfe von Urinproben nachgewiesen werden, deren Empfindlichkeit bei Primärtumoren alles andere als zufriedenstellend war (60 %), deren Nachweis jedoch möglich ist können durch die leichte Abbaubarkeit von RNA in der normalen Umgebung beeinträchtigt werden11.Genetische und epigenetische Marker stehen beide vor dem Problem der Prävalenz in Tumoren und der Beschränkung auf Krebsarten.
Die DNA-Methylierung ist ein starker Kandidat als Biomarker für die Früherkennung, seit sie 1983 von Feinberg erstmals mit Krebs in Verbindung gebracht wurde12. Abweichungen der DNA-Methylierung werden in allen Krebsstadien beobachtet, bereits im präkanzerösen Stadium.Eine abweichende DNA-Hypermethylierung findet normalerweise auf CpG-Inseln in Genpromotoren statt, um Tumorsuppressoren entgegenzuwirken13,14.Studien deuten auch darauf hin, dass eine abnormale DNA-Hypermethylierung zur Hochregulierung von Entwicklungsregulatoren führt15.Das DNA-Methylierungstal, das häufig mit Entwicklungsregulatoren und hypermethylierten Krebsarten in Verbindung gebracht wird, könnte den Genexpressionsmodus auf einen stabileren DNA-Methylierung-abhängigen Modus umstellen und die Verbindung zu methyliertem Histon H3K27me3 und assoziierten Polycomb-Proteinen verringern16,17.
Unter der großen Anzahl veröffentlichter DNA-Methylierungsmarker haben mehrere erfolgreich auf dem Markt eingeführt;Allerdings schöpfen die derzeit kommerzialisierten DNA-Methylierungsmarker und Diagnosepanels aus mehreren Gründen das Potenzial der Krebsfrüherkennung noch nicht vollständig aus18.Während diese Biomarker unter Verwendung von Datenbankinformationen meist eine akzeptable Leistung zeigen, erbringen sie in der realen Welt in der Regel weniger optimale Ergebnisse, da reale Proben oft komplexer und nicht so repräsentativ sind wie die in den Datenbanken ausgewählten.Es wurde gezeigt, dass die auf Next-Generation-Sequencing basierende Früherkennung mit Methylierung mehrerer Krebsarten bei Krebserkrankungen im Stadium I und II lediglich eine Sensitivität von 16,8 % bzw. 40,4 % aufweist19.Früherkennungstests erfordern eine größere Stabilität und genauere Biomarker.
Entdeckung universeller Krebsmarker (UCOM) mithilfe von Guide Positioning Sequencing (GPS)
Trotz jahrzehntelanger Krebsforschung konnten keine zufriedenstellende Prävention und Behandlung erzielt werden.Es sind neue Methoden erforderlich, um Forschern eine gründliche Beurteilung von Krebs zu ermöglichen.In den letzten 23 Jahren wurden sechs Krebsmerkmale wie die Vermeidung von Apoptose, Gewebeinvasion und -metastasierung usw. auf 14 erweitert, indem Merkmale wie nichtmutationsbedingte epigenetische Reprogrammierung und polymorphe Mikrobiome einbezogen wurden20,21.Je mehr Details über Krebs bekannt werden, desto mehr Perspektiven eröffnen sich für die Krebsforschung.Die Krebsforschung ist in zwei Richtungen (Gemeinsamkeit und Individualität) allmählich in ein neues Zeitalter eingetreten.Mit der Entwicklung der Präzisionsonkologie in den letzten Jahren verlagert sich der Fokus der Krebsforschung auf die individualisierte zielgerichtete Therapie und die Heterogenität von Krebs22.Daher konzentrierten sich kürzlich identifizierte Krebsbiomarker hauptsächlich auf bestimmte Krebsarten, wie z. B. PAX6-Zervixkarzinom23 und BMP3 für Darmkrebs24.Die Leistung dieser krebsartenspezifischen Biomarker schwankt, dennoch ist es aufgrund der begrenzten biologischen Probenentnahme und der hohen Kosten für anfällige Personen immer noch nicht möglich, sich einem Screening auf alle Krebsarten gleichzeitig zu unterziehen.Es wäre ideal, wenn wir einen einzigen, robusten Biomarker identifizieren könnten, der für alle Krebsarten in einem frühen Stadium wirksam ist.
Um solch ein ideales Ziel zu erreichen, muss ein besserer Biomarker-Kandidat aus der Liste potenzieller Biomarker-Typen ausgewählt werden.Es ist bekannt, dass DNA-Methylierungsfehler unter allen genetischen und epigenetischen Profilen mit Krebs in Zusammenhang stehen und zu den frühesten, wenn nicht ersten, krebsbedingten Anomalien gehören, die chronologisch auftreten.Die Untersuchung der DNA-Methylierung begann früh, wurde jedoch durch den Mangel an Forschungsmethoden behindert.Unter den 28 Millionen potenziell methylierten CpG-Stellen im Genom muss eine überschaubare Anzahl erkannt und dem Genom zugeordnet werden, um die Tumorentstehung besser zu verstehen.Die Bisulfit-Sequenzierung des gesamten Genoms (WGBS), die als Goldstandard der DNA-Methylierungssequenzierung gilt, kann nur 50 % der Cs in Krebszellen abdecken, da die Bisulfitbehandlung DNA-Fragmente aufbricht und dabei die Komplexität des Genoms verringert die Umwandlung von Cs-zu-Ts25.Andere Methoden wie 450k-Chips decken nur 1,6 % der Genommethylierung ab.Basierend auf 450.000 Daten weist ein DNA-Methylierungs-Nachweispanel eine Sensitivität von 35,4 % für 6 Krebsarten im Stadium I auf26.Beschränkungen der Krebsarten, schlechte Leistung und Rauschen, das durch Erkennungsmethoden im Analyseprozess entsteht, sind zu den größten Hindernissen für Pan-Krebs-Erkennungspanels geworden.
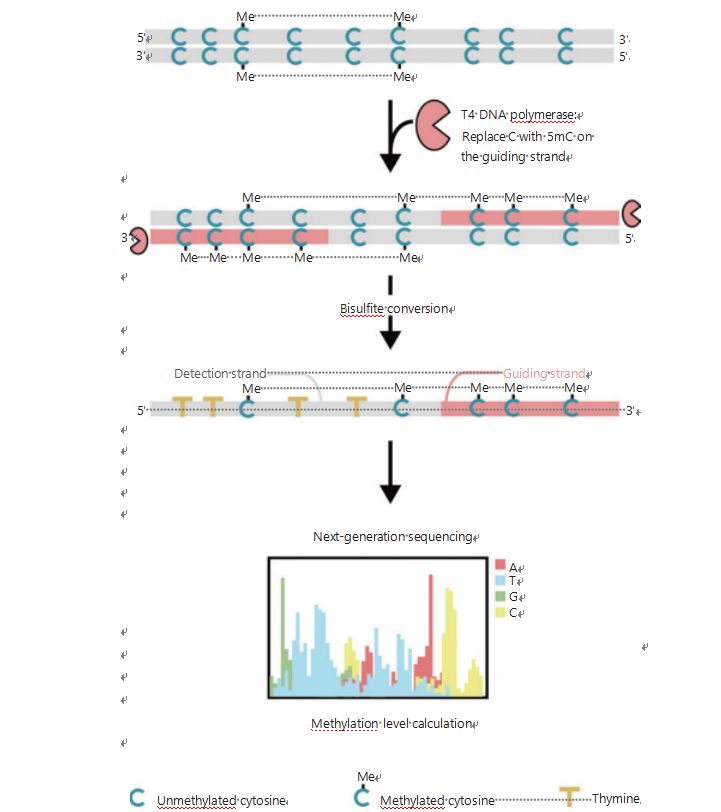
Um die epigenetischen Muster von Zellen während der Tumorentstehung und Metastasierung besser untersuchen zu können, haben wir ein einzigartiges GPS für die genomweite DNA-Methylierungserkennung entwickelt, das bis zu 96 % der CpG-Stellen in 0,4 Milliarden Lesevorgängen abdeckt25.GPS ist eine bilaterale Sequenzierungsmethode, die ein 3‘-Ende eines DNA-Fragments von nicht umwandelbaren Methylcytosinen nach Bisulfitbehandlung verwendet und die Ausrichtung der DNA-Methylierungsberechnung des 5‘-Endes durch Paarendsequenzierung steuert (Abbildung 1)25.Der Methyl-Cytosin-Leitstrang, der als Matrizenstrang fungiert, hilft bei der Ausrichtung der Region mit hohem GC-Wert, wodurch die am häufigsten aufgegebenen Sequenzierungsdaten in herkömmlichem WGBS wiederhergestellt werden.Die hohe Abdeckungsfunktion von GPS liefert eine enorme Menge an DNA-Methylierungsinformationen, die es uns ermöglicht, Krebsmethylierungsprofile in bisher wenig untersuchten Regionen mit einer erheblich höheren Auflösung zu untersuchen.
GPS bietet uns ein leistungsstarkes Werkzeug zur Untersuchung der Homogenität von Krebs, das die Krebsforschung erheblich vereinfachen und möglicherweise eine universelle Erklärung für Tumorentstehung und Metastasierung finden kann.Bei der Analyse von GPS-Daten von Krebszelllinien trat häufig ein einzigartiges Phänomen auf.Es gab eine Reihe von Regionen, die in mehreren Krebsproben ungewöhnlich hypermethyliert zu sein schienen.Dieser unerwartete Befund wurde anschließend als UCOM validiert.Mehr als 7.000 Proben von 17 Krebsarten in der Datenbank The Cancer Genome Atlas (TCGA) wurden analysiert. Darunter identifizierten wir das erste UCOM, HIST1H4F, ein Histon-verwandtes Gen, das bei allen Krebsarten hypermethyliert ist27.Anschließend wurde eine Reihe von UCOMs in der TCGA-Datenbank, der Gene Expression Omnibus (GEO)-Datenbank und in klinischen Proben aus der Praxis gefunden und validiert.Bisher wurden HIST1H4F, PCDHGB7 und SIX6 als UCOMs gefunden und validiert.Die unerwartete Entdeckung von UCOMs bietet eine wirkungsvolle Antwort auf die Notwendigkeit einer Früherkennung von Krebs.UCOMs bieten eine Lösung für die Einzelmarkererkennung mehrerer Krebsarten.
Eigenschaften von UCOMs
Bei der Validierung wurde gezeigt, dass UCOMs vier Hauptmerkmale aufweisen, die es UCOMs ermöglichen, die Wirksamkeit aktueller Biomarker zu übertreffen (Abbildung 2).
Einzigartig bei bösartigen Erkrankungen
UCOMs treten nur bei krebsartigen oder präkanzerösen Läsionen auf und werden von normalen physiologischen Veränderungen nicht beeinflusst.Einige der aktuellen krebsbezogenen Marker, die häufig in der Früherkennung und/oder beim Screening eingesetzt werden, haben zu Überdiagnosen geführt.Erhöhte PSA-Werte, ein klinisch anerkanntes Screening-Instrument, werden auch bei gutartigen Erkrankungen wie Prostatahyperplasie und Prostatitis festgestellt10.Die Überdiagnose und die daraus resultierende Überbehandlung führen zu einer verminderten Lebensqualität aufgrund von Darm-, Harn- und Sexualkomplikationen28.Andere proteinbasierte und im klinischen Umfeld weit verbreitete Biomarker wie CA-125 haben keine signifikanten Vorteile gebracht und gleichzeitig zu Überdiagnosen und Überbehandlungen geführt29.Die hohe Spezifität von UCOMs für maligne Erkrankungen vermeidet diese Mängel.Das UCOM, PCDHGB7, unterscheidet effizient hochgradige Plattenepithelläsionen (HSILs) und Gebärmutterhalskrebs von normalen Proben und niedriggradige Plattenepithelläsionen (LSILs), während die meisten anderen Biomarker nur Gebärmutterhalskrebs von normalen Proben unterscheiden können30.Obwohl PCDHGB7 keine signifikanten Unterschiede zwischen normalem Endometrium und Endometriumhyperplasie erkennt, werden signifikante Unterschiede zwischen normalem Endometrium und atypischer Hyperplasie festgestellt, und basierend auf PCDHGB731 werden noch größere Unterschiede zwischen normalem Endometrium und Endometriumkrebs (EC) festgestellt.UCOMs gelten nur für bösartige Läsionen in Datenbanken und klinischen Proben.Aus der Sicht des Patienten verringern einzigartige UCOMs die Schwelle zum Verständnis komplexer Indikationen verschiedener instabiler Biomarker mit schlechter Leistung und die entsprechende Angst während des Bewertungsprozesses.Aus Sicht des Arztes unterscheiden einzigartige UCOMs bösartige von gutartigen Läsionen, was die Triage von Patienten erleichtert und unnötige medizinische Eingriffe und Überbehandlungen reduziert.Daher reduzieren einzigartige UCOMs die Redundanz des medizinischen Systems, lindern die Systembelastung und stellen den Bedürftigen mehr medizinische Ressourcen zur Verfügung.
Abbildung 1 Schematische Darstellung des GPS-Arbeitsablaufs zur DNA-Methylierungserkennung25.Graue Linie: eingegebene DNA-Sequenz;rote Linie: Mit T4-DNA-Polymerase behandelte DNA, die Cytosin durch 5′-Methylcytosin am 3′-Ende der Eingabe ersetzt;blaues C mit Me: methyliertes Cytosin;blau C: unmethyliertes Cytosin;gelbes T: Thymin25.
Alles oder nichts
UCOMs kommen nur in Krebszellen vor und werden in fast allen Krebszellen stabil nachgewiesen.Es wurde bestätigt, dass HIST1H4F in fast allen Tumorarten hypermethyliert ist, jedoch nicht in normalen Proben27.Ebenso wurde gezeigt, dass PCDHGB7 und SIX6 in allen Tumorproben hypermethyliert sind, jedoch nicht in normalen Proben30-32.Diese einzigartige Eigenschaft verbessert die Leistung von UCOMs hinsichtlich der Nachweisgrenze und Empfindlichkeit erheblich.Nur 2 % der Krebszellen können in Proben differenziert werden, was UCOMs zu einem viel empfindlicheren Biomarker macht als die meisten vorhandenen Biomarker30. Als Biomarker zur Erkennung von Darmkrebs kommen KRAS-Mutationen nur in etwa 36 % der Fälle von Darmkrebs vor. was auf ein schlechtes diagnostisches Potenzial hindeutet33.Die geringe Prävalenz von KRAS-Mutationen bei Darmkrebs schränkt die Kombination von KRAS mit anderen Biomarkern ein.Tatsächlich mag eine Kombination von Biomarkern auf den ersten Blick vielversprechend erscheinen, führt jedoch nicht immer zu einem zufriedenstellenden Ergebnis, weist jedoch ein viel größeres Rauschen in der Nachweisanalyse auf und erfordert in der Regel kompliziertere experimentelle Verfahren.Im Gegensatz dazu kommen PCDHGB7 und andere UCOMs bei allen Krebsarten vor.UCOMs erkennen krebserregende Bestandteile in verschiedenen Arten von Krebsproben mit höchster Präzision und machen gleichzeitig komplexe Analyseprozesse mit Rauschunterdrückung überflüssig.Es ist nicht schwer, Krebs in einer großen Probe zu erkennen, aber es ist äußerst schwierig, Krebs in einer kleinen Probe zu erkennen.UCOMs sind in der Lage, kleine Mengen Krebs zu erkennen.
Abbildung 2 Eigenschaften von UCOMs.
Krebserkennung vor pathologischen Veränderungen
UCOMs können im präkanzerösen Stadium vor pathologischen Veränderungen nachgewiesen werden.Als epigenetische Biomarker treten UCOM-Anomalien in einem früheren Stadium auf als phänotypische Anomalien und sind während der gesamten Tumorentstehung, -progression und -metastasierung nachweisbar34,35.Die Empfindlichkeit von UCOM im Laufe der Zeit verbessert die Leistung von UCOM bei der Erkennung von Krebs im Frühstadium und präkanzerösen Läsionen.Die Erkennung von Krebs im Frühstadium anhand von Biopsien und Zytologie kann selbst für die erfahrensten Pathologen schwierig sein.Eine einzelne mittels Kolposkopie gewonnene Biopsie wurde in 60,6 % der HSIL+-Proben als positiv gemeldet.Bei mehreren Läsionen sind zusätzliche Biopsien erforderlich, um die Empfindlichkeit zu erhöhen36.Im Gegensatz dazu weist das UCOM, PCDHGB7, eine Sensitivität von 82 % für HSIL+-Proben auf und übertrifft damit die Sensitivität von Biopsien und den meisten Biomarkern30.Der Methylierungsmarker FAM19A4 hat eine Sensitivität von 69 % für CIN2+, was der Zytologie ähnelt, CIN1 jedoch nicht von normalen Proben unterscheiden kann37.Es hat sich gezeigt, dass UCOMs ein viel empfindlicherer Biomarker für die Früherkennung sind.Im Vergleich zu erfahrungsbasierten Pathologen weisen UCOMs eine überlegene Erkennungsempfindlichkeit für Krebserkrankungen im Frühstadium auf, was wiederum zu einer verbesserten Krebsprognose und einem besseren Überleben beiträgt30.Darüber hinaus bieten UCOMs eine Erkennungsplattform, die auch für Bereiche zugänglich ist, in denen es an erfahrenen Pathologen mangelt, und die die Erkennungseffizienz erheblich verbessert.Mit einheitlichen Probenahme- und Nachweisverfahren liefert die UCOM-Erkennung stabile und leicht zu interpretierende Ergebnisse, die besser zu einem Screening-Protokoll passen, das weniger Fachpersonal und medizinische Ressourcen erfordert.
Leicht zu erkennen
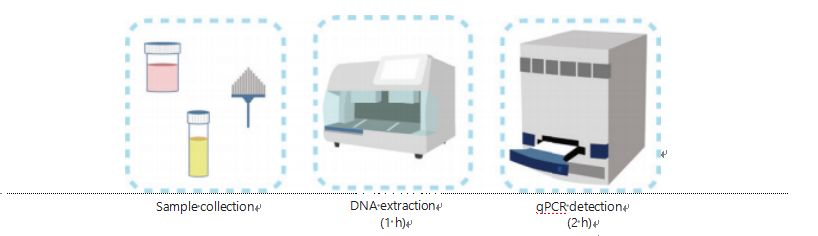
Aktuelle Methoden zum Nachweis der DNA-Methylierung sind kompliziert und zeitaufwändig.Die meisten Methoden erfordern eine Bisulfit-Umwandlung, was zu einem Verlust der Probenqualität und möglicherweise zu instabilen und ungenauen Ergebnissen führt.Die durch die Bisulfitbehandlung verursachte schlechte Reproduzierbarkeit führt möglicherweise zu Verwirrung bei Ärzten und Patienten und beeinträchtigt die Nachsorge- und/oder Behandlungsstrategien zusätzlich.Daher haben wir die Methode des UCOM-Nachweises weiter modifiziert, um eine problematische Bisulfitbehandlung der Proben zu vermeiden, den Anforderungen der klinischen Anwendung gerecht zu werden und die Zugänglichkeit zu verbessern.Wir haben eine neuartige Methode entwickelt, die methylierungsempfindliche Restriktionsenzyme in Kombination mit quantitativer Echtzeit-Fluoreszenz-PCR (Me-qPCR) verwendet, um den Methylierungsstatus von UCOMs innerhalb von 3 Stunden unter Verwendung einfach zu handhabender Verfahren zu quantifizieren (Abbildung 3).Me-qPCR kann mehrere Probentypen umfassen, wie z. B. die klinische Sammlung von Körperflüssigkeiten und selbst gesammelte Urinproben.Gesammelte klinische Proben können verarbeitet, gelagert und mithilfe einer standardisierten und automatisierten DNA-Extraktion problemlos zum Nachweis weitergeleitet werden.Die extrahierte DNA kann dann direkt auf die Me-qPCR-Plattform angewendet werden, um eine Eintopfreaktion durchzuführen und Quantifizierungsergebnisse auszugeben.Nach einer einfachen Ergebnisanalyse unter Verwendung diagnostischer Modelle, die an bestimmte Krebsarten angepasst und validiert wurden, wird die endgültige Bestimmung der UCOM-Erkennungsergebnisse interpretiert und als halbquantitativer Wert dargestellt.Die Me-qPCR-Plattform übertrifft die herkömmliche Bisulfit-Pyrosequenzierung beim UCOM-Nachweis und spart gleichzeitig 3 Stunden Bisulfit-Umwandlung, gemäß dem EZ DNA Methylation-Gold-Kit-Protokoll.Die innovative Methylierungserkennungsplattform macht die UCOM-Erkennung stabiler, genauer und zugänglicher30.
Abbildung 3 Erkennungsprozess von UCOMs.Zu den Probentypen gehören professionell entnommener BALF, Pap-Bürste und/oder selbst gesammelter Urin.Der DNA-Extraktionsprozess kann in einem automatischen Extraktor untergebracht werden, dessen Produkt direkt durch qPCR nachgewiesen werden kann.
Anwendung von UCOMs
Lungenkrebs
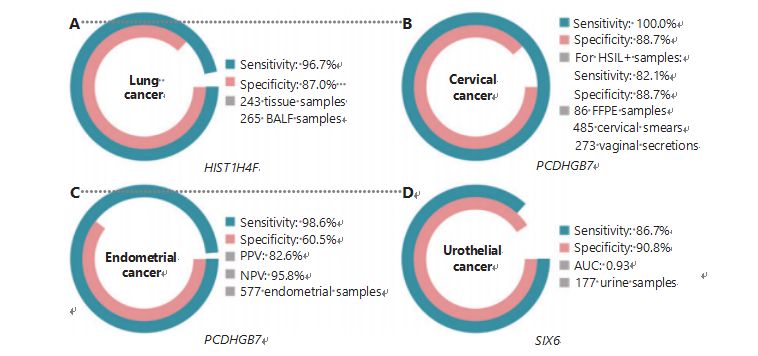
Lungenkrebs ist die am zweithäufigsten diagnostizierte und tödlichste Krebsart weltweit und macht 11,4 % der Neuerkrankungen und 18,0 % der neuen Todesfälle aus1.Von allen Diagnosen handelt es sich bei 85 % um nicht-kleinzelligen Lungenkrebs (NSCLC) und bei 15 % um kleinzelligen Lungenkrebs (SCLC), der einen höheren Grad an Bösartigkeit aufweist38.Die niedrig dosierte Computertomographie (LDCT) ist die derzeit empfohlene Screening-Methode für Lungenkrebs und verbessert nachweislich die Früherkennung und senkt die Mortalität6;Aufgrund der geringen Spezifität und der schlechten Zugänglichkeit hat sich die LDCT jedoch noch nicht als zufriedenstellende Screening-Methode bewährt, ebenso wie andere häufige Krebsmarker wie CEA39.Die Kosten und das Potenzial für verpasste Diagnosen und Fehldiagnosen der LDCT-Screening-Strategie behindern den Fortschritt der Förderung der Lungenkrebs-Screening40.HIST1H4F, ein UCOM, hat ein enormes Potenzial als Früherkennungs-Biomarker in Proben der bronchoalveolären Flüssigkeit (BALF)27.HIST1H4F ist bei Lungenadenokarzinomen und Plattenepithelkarzinomen der Lunge hypermethyliert, mit einer Nachweisspezifität von 96,7 % und einer Sensitivität von 87,0 % (Abbildung 4A) und einer außergewöhnlichen Leistung bei Krebserkrankungen im Stadium I27.HIST1H4F hat eine Spezifität von 96,5 % und eine Sensitivität von 85,4 % für NSCLC und 96,5 % bzw. 95,7 % für SCLC27.Darüber hinaus haben Proben von acht anderen Krebsarten, darunter Bauchspeicheldrüsen- und Darmkrebs, bestätigt, dass HIST1H4F bei allen acht Krebsarten hypermethyliert ist27.
Gebärmutterhalskrebs
Gebärmutterhalskrebs war im Jahr 2020 die am vierthäufigsten diagnostizierte Krebsart und die vierthäufigste Krebstodesursache bei Frauen und machte weltweit 3,1 % der Neuerkrankungen und 3,4 % der krebsbedingten Todesfälle aus1.Um Gebärmutterhalskrebs bis 2030 zu eliminieren, wie von der WHO vorgeschlagen, ist die Früherkennung von Gebärmutterhalskrebs eine Notwendigkeit.Bei frühzeitiger Erkennung beträgt die 5-Jahres-Überlebensrate bei invasivem Gebärmutterhalskrebs 92 %41.Die Richtlinien der American Cancer Society (ACS) empfehlen zervikale Zytologietests, primäre HPV-Tests oder Co-Tests für das Screening42.Die Zervixzytologie ist invasiv und kann nur 63,5 % der CIN2+-Fälle erkennen37.
Im Gegensatz dazu hat PCDHGB7 bei der Verwendung von Pap-Abstrichen und Vaginalsekret viel bessere Ergebnisse erzielt und kann HSIL in einem ultrafrühen Stadium effizient von LSIL unterscheiden.PCDHGB7 allein hat eine Sensitivität von 100,0 % und eine Spezifität von 88,7 % für Gebärmutterhalskrebs (Abbildung 4B) und eine Sensitivität von 82,1 % und eine Spezifität von 88,7 % für HSIL+-Proben30.PCDHGB7 weist außerdem eine Sensitivität von 90,9 % und eine Spezifität von 90,4 % in Vaginalsekretproben für Gebärmutterhalskrebs auf, die viel einfacher zu sammeln sind30.In Kombination mit dem Hochrisiko-(hr)HPV-Test oder dem Thinprep Cytology Test (TCT) weist PCDHGB7 eine erhöhte Sensitivität von 95,7 % und eine Spezifität von 96,2 % auf und übertrifft damit deutlich die des hrHPV-Tests (20,3 %), TCT (51,2 %). ) und beide zusammen (57,8 %) bei Gebärmutterhalskrebs30.Es wurde auch gezeigt, dass PCDHGB7 bei 17 Krebsarten aus der TCGA-Datenbank hypermethyliert ist, was auf seine Eignung für die UCOM-Familie hinweist30.
Abbildung 4 UCOMs wurden in groß angelegten klinischen Studien bei vier Krebsarten validiert.A. Leistung von HIST1H4F, einem UCOM, bei der Lungenkrebserkennung von 508 Proben.B. Leistung von PCDHGB7, einem UCOM, bei der Erkennung von Gebärmutterhalskrebs in 844 Proben.C. Leistung von PCDHGB7, einem UCOM, bei der Endometriumkrebserkennung von 577 Endometrium-Pap- und Tao-Bürstenproben.D. Leistung von SIX6, einem UCOM, bei der Erkennung von Urothelkrebs in 177 Proben.
EC
EC ist eine der häufigsten Krebserkrankungen des weiblichen Fortpflanzungssystems weltweit, mit geschätzten 4,2 Millionen Neuerkrankungen und 1 % der krebsbedingten Todesfälle pro Jahr1.Bei erfolgreicher Diagnose in einem frühen Stadium ist EC heilbar und hat eine 5-Jahres-Überlebensrate von 95 % bei Krebs im Stadium I.Patientinnen mit Symptomen, wie z. B. abnormalen Uterusblutungen, werden regelmäßig klinisch untersucht und invasiven und schmerzhaften Biopsieverfahren unterzogen, obwohl nur 5–10 % letztendlich EC43 entwickeln.Der transvaginale Ultraschall als übliche Nachweismethode ist äußerst unzuverlässig, da er nicht in der Lage ist, gutartige von bösartigen Endometriumveränderungen zu unterscheiden, und die hohe Falsch-Positiv-Rate44.
Es wurde ein paralleler Vergleich von Serum-CA-125, einem weit verbreiteten EC-Biomarker, und PCDHGB7 durchgeführt.Serum-CA-125 hatte eine Sensitivität von 24,8 %, was darauf hindeutet, dass CA-125 trotz einer Spezifität von 92,3 % ein unzureichender Marker für EC ist31.Der PCDHGB7-Nachweis mit Pap-Bürstenproben ergab eine Sensitivität von 80,65 % und eine Spezifität von 82,81 % für ECatall-Stadien, während ein Tao-Bürste eine Sensitivität von 61,29 % und eine Spezifität von 95,31 % aufwies31.Das PCDHGB7-Diagnosemodell, basierend auf Me-qPCR, ergab eine Sensitivität von 98,61 %, eine Spezifität von 60,5 % und eine Gesamtgenauigkeit von 85,5 % unter Verwendung von Pap- und Tao-Pinselproben (Abbildung 4C)31.
Urothelkrebs
Urothelkrebs, bestehend aus Blasen-, Nierenbecken- und Harnleiterkrebs, war im Jahr 2020 weltweit die siebthäufigste Krebserkrankung und verursachte 5,2 % der Neuerkrankungen und 3,9 % der Todesfälle1.Urothelkarzinome, von denen mehr als 50 % Blasenkrebs sind, waren im Jahr 2022 die vierthäufigste Krebsdiagnose in den Vereinigten Staaten und machten 11,6 % der neu diagnostizierten Fälle aus3.Ungefähr 75 % der Blasenkrebserkrankungen werden als nicht muskelinvasiver Blasenkrebs klassifiziert, der auf die Schleimhaut oder Submukosa beschränkt ist45.Eine Zystoskopiebiopsie ist der Goldstandard für die Diagnose von Urothelkrebs, durchgeführt durch Fluoreszenz-in-situ-Hybridisierung (FISH) und zytologische Tests.FISH und Zytologie weisen eine schlechte diagnostische Leistung auf, und die Zystoskopie ist ein Eingriff und birgt das Risiko, Mikroläsionen zu übersehen, Läsionen falsch zu interpretieren und möglicherweise eine Ausbreitung oder einen Rückfall von Krebs zu verursachen46.Es wurde auch gezeigt, dass das zuvor validierte UCOM, PCDHGB7, bei Urothelkrebs hypermethyliert ist, mit einer Fläche unter der Kurve von 0,86, was auf eine potenzielle diagnostische Fähigkeit hindeutet30.Um mehr UCOMs weiter zu validieren und mehr Probentypen besser unterzubringen, wurde SIX6, ein neuartiges UCOM, untersucht und zeigte ein hervorragendes diagnostisches Potenzial bei der Früherkennung von Urothelkrebs mithilfe von Urinproben auf der Me-qPCR-Plattform.Der SIX6-Nachweis anhand von Urinproben zeigte eine kompetitive Sensitivität von 86,7 % und eine Spezifität von 90,8 % (Abbildung 4D) und war gleichzeitig nicht-invasiv und einfach zu erfassen32.Das Potenzial von SIX6 bei der Metastasenüberwachung und der Bewertung der Behandlungswirksamkeit wird derzeit untersucht.
Die Zukunft und Herausforderungen
UCOMs weisen ein starkes diagnostisches Potenzial für mehrere Krebsarten auf, es gibt jedoch noch viel zu tun.Wir haben die Liste der UCOMs erweitert und UCOMs aktiv bei weiteren Krebsarten validiert, darunter auch solchen, die traditionell schwer zu erkennen sind.Validierungsergebnisse aus TCGA-Datenbanken haben die Anwendung von UCOMs bei mehr Krebsarten und mehr Situationen weiter bestätigt.In einer vorläufigen Untersuchung wurde gezeigt, dass UCOMs ein robustes diagnostisches Potenzial für Cholangiokarzinome und Pankreas-Adenokarzinome haben, die mit aktuellen Screening-Methoden in einem frühen Stadium kaum zu diagnostizieren sind32,47.Die Fähigkeit, seltene Krebsarten mit UCOMs zu erkennen, kann durch eine verbesserte Flüssigbiopsieplattform mit zirkulierender Tumor-DNA (ctDNA) genutzt werden48.Eine Studie mit einem Plasma-DNA-basierten Pan-Krebs-Erkennungspanel ergab eine Sensitivität von 57,9 %49.Trotz der hohen Spezifität zeigt die Gesamtleistung, dass es noch Raum für Verbesserungen gibt.
Die einzigartigen Eigenschaften von UCOMs haben auch die Untersuchung des UCOM-Potenzials bei der Bewertung der Behandlungswirksamkeit und der Wiederholungsüberwachung unterstützt.Gemäß den Response Evaluation Criteria in Solid Tumors (RECIST) ist die medizinische Bildgebung die empfohlene Methode zur Rezidivüberwachung und Bewertung der Behandlungswirksamkeit, während Tumormarker allein zur Beurteilung verwendet werden50.In der Realität werden bildgebende Verfahren jedoch stark von der Häufigkeit und dem Zeitpunkt beeinflusst und setzen Patienten daher einem höheren Risiko und höheren Kosten aus51,52.SIX6 wurde als Prädiktor für die Metastasierung von Brustkrebs validiert32.Die auf Flüssigbiopsie basierende ctDNA-Überwachung ermöglicht eine Echtzeitüberwachung über minimale Resterkrankungen Monate vor der radiologischen Erkennung und verzögert und verhindert im Idealfall das Fortschreiten des rezidivbedingten Krebses53.Vorläufige Ergebnisse deuten darauf hin, dass UCOMs den Grad der krebsartigen Hypermethylierung in Echtzeit unmittelbar nach der Operation und Behandlung widerspiegeln32.Die hohe Empfindlichkeit von UCOMs und die Anwendbarkeit auf mehrere nicht-intrusive Probentypen ermöglichen es UCOMs, als präziser Rezidiv-Monitoring-Biomarker zu dienen und gleichzeitig eine hohe Patientencompliance aufrechtzuerhalten.
Gleichzeitig ist die öffentliche Zugänglichkeit des Tests ein weiteres großes Thema, das zusätzlichen Aufwand erfordert.Während UCOM-Erkennungskooperationen in immer mehr Krankenhäusern eingeführt wurden, in der Hoffnung, mehr Patienten davon zu profitieren, wurden in ländlichen Gebieten Chinas aktiv kostenlose Erkennungen und Untersuchungen durchgeführt.UCOMs erfordern eine verbesserte Zugänglichkeit, um sich als praktikables Screening-Instrument zu qualifizieren, insbesondere für unterentwickelte Gebiete.
Während die Ergebnisse der UCOM-Anwendung bei der Früherkennung vielversprechend sind, gibt es viele Unbekannte über UCOM.Bei aktiver Erforschung sind weitere Untersuchungen darüber erforderlich, warum UCOMs bei Krebserkrankungen überall vorkommen.Die zugrunde liegenden epigenetischen Regulationsmechanismen, die UCOMs zugrunde liegen, sind einer weiteren Untersuchung wert, was eine neue Richtung für Krebstherapeutika rechtfertigen könnte.Um auf das Zusammenspiel zwischen Tumorhomogenität und -heterogenität zurückzukommen, interessiert uns die Frage, warum UCOMs eine Ausnahme von der Mehrzahl der Krebsbiomarker sein können, die eng mit bestimmten Krebsarten verknüpft sind.Die Rolle von UCOM-identifizierten DNA-Methylierungsfehlern bei der Tumorentstehung, Tumorprogression und Metastasierung wurde im Prozess des Verlusts und der Wiederherstellung der Zellidentität nicht geklärt und erfordert eine gründliche Untersuchung.Ein weiteres großes Interesse liegt im Bereich der Integration des Homogenitätsmerkmals von UCOMs mit gewebespezifischen Markern in der Hoffnung, auf umgekehrte Weise eine präzise Erkennung von Krebsspuren und die Identifizierung von Tumorgewebeursprüngen zu erreichen.UCOMs können ein ideales Instrument sein, um Krebs vorzubeugen, Krebs zu erkennen und möglicherweise Krebs zu verteidigen und zu beseitigen.
Gewähren Sie Unterstützung
Diese Arbeit wurde vom National Key R&D Program of China (Grant No. 2022BEG01003), der National Natural Science Foundation of China (Grant Nos. 32270645 und 32000505) und einem Grant der Heilongjiang Provincial Health Commission (Grant No. 2020-111) unterstützt. und ein Stipendium des Heze Science and Technology Institute (Stipendium Nr. 2021KJPT07).
Erklärung zum Interessenkonflikt
Wei Li ist F&E-Direktor für Shanghai Epiprobe Biotechnology Co., Ltd. Wenqiang Yu ist Mitglied des wissenschaftlichen Beirats von Epiprobe.W. Yu und Epiprobe haben angemeldete Patente im Zusammenhang mit dieser Arbeit genehmigt.Alle anderen Autoren erklären keine Interessenkonflikte.
Autorenbeiträge
Konzipiert und gestaltet das Projekt: Chengchen Qian und Wenqiang Yu.
Verfasser des Artikels: Chengchen Qian.
Die Illustrationen stammen von Chengchen Qian.
Das Manuskript überprüft und bearbeitet: Xiaolong Zou, Wei Li, Yinshan Li und Wenqiang Yu.
Verweise
1. Sung H, Ferlay J, Siegel RL, LaversanneM, Soerjomataram I, Jemal A, et al.Globale Krebsstatistik 2020: GLOBOCAN-Schätzungen von
Inzidenz und Mortalität weltweit für 36 Krebsarten in 185 Ländern.CA Cancer J Clin.2021;71: 209-49.
2. Xia C, Dong X, Li H, Cao M, Sun D, He S, et al.Krebsstatistiken in China und den Vereinigten Staaten, 2022: Profile, Trends und Determinanten.Chin MedJ (Engl.).2022;135: 584-90.
3. Siegel RL, Miller KD, WagleNS, JemalA.Krebsstatistik, 2023. CA Cancer J Clin.2023;73: 17-48.
4. Crosby D, BhatiaS, Brindle KM, Coussens LM, Dive C, Emberton M, et al.Früherkennung von Krebs.Wissenschaft.2022;375: eaay9040.
5. Ladabaum U, Dominitz JA, KahiC, Schoen RE.Strategien für
Darmkrebs-Screening.Gastroenterologie.2020;158: 418-32.
6. Tanoue LT, Tanner NT, Gould MK, Silvestri GA.Lungenkrebs-Screening.Am J Respir Crit Care Med.2015;191: 19-33.
7. Bouvard V, WentzensenN, Mackie A, Berkhof J, BrothertonJ, Giorgi-Rossi P, et al.Die IARC-Perspektive zur Früherkennung von Gebärmutterhalskrebs.N EnglJ Med.2021;385: 1908-18.
8. Xue P, Ng MTA, QiaoY.Die Herausforderungen der Kolposkopie für die Gebärmutterhalskrebsvorsorge in LMICs und Lösungen durch künstliche Intelligenz.BMC Med.2020;18: 169.
9. Johnson P, Zhou Q, Dao DY, Lo YMD.Zirkulierende Biomarker bei der Diagnose und Behandlung von hepatozellulärem Karzinom.Nat Rev Gastroenterol Hepatol.2022;19: 670-81.
10. Van PoppelH, Albreht T, Basu P, HogenhoutR, CollenS, Roobol M. Serum-PSA-basierte Früherkennung von Prostatakrebs in Europa und weltweit: Vergangenheit, Gegenwart und Zukunft.Nat Rev Urol.2022;19:
562-72.
11. HolyoakeA, O'Sullivan P, Pollock R, Best T, Watanabe J, KajitaY,
et al.Entwicklung eines Multiplex-RNA-Urintests zur Erkennung und Stratifizierung von Übergangszellkarzinomen der Blase.Clin Cancer Res.2008;14: 742-9.
12. Feinberg AP, Vogelstein B. Hypomethylierung unterscheidet Gene einiger menschlicher Krebsarten von ihren normalen Gegenstücken.Natur.1983;301: 89-92.
13. Ng JM, Yu J. Promotor-Hypermethylierung von Tumorsuppressorgenen als potenzielle Biomarker bei Darmkrebs.IntJ Mol Sci.2015;16: 2472-96.
14. Esteller M. Krebsepigenomik: DNA-Methylome und Histon-Modifikationskarten.Nat Rev Genet.2007;8: 286-98.
15. Nishiyama A, Nakanishi M. Navigieren in der DNA-Methylierungslandschaft von Krebs.Trends Genet.2021;37: 1012-27.
16. Xie W, Schultz MD, ListerR, Hou Z, Rajagopal N, Ray P, et al.Epigenomische Analyse der Multilinien-Differenzierung menschlicher embryonaler Stammzellen.Zelle.2013;153: 1134-48.
17. Li Y, Zheng H, Wang Q, Zhou C, WeiL, Liu X, et al.Genomweite Analysen zeigen, dass Polycomb eine Rolle bei der Förderung der Hypomethylierung von DNA-Methylierungstälern spielt.Genombiol.2018;19:18.
18. Koch A, JoostenSC, Feng Z, de Ruijter TC, DrahtMX, MelotteV,
et al.Analyse der DNA-Methylierung bei Krebs: Standort erneut besucht.Nat Rev Clin Oncol.2018;15: 459-66.
19. KleineEA, Richards D, Cohn A, TummalaM, Lapham R, Cosgrove D, et al.Klinische Validierung eines gezielten, auf Methylierung basierenden Früherkennungstests für mehrere Krebsarten unter Verwendung eines unabhängigen Validierungssatzes.Ann Oncol.2021;32: 1167-77.
20. Hanahan D, Weinberg RA.Die Kennzeichen von Krebs.Zelle.2000;100: 57-70.
21. Hanahan D. Merkmale von Krebs: neue Dimensionen.Krebsentdeckung.2022;12: 31-46.
22. Schwartzberg L, Kim ES, Liu D, Schrag D. Präzisionsonkologie: Wer, wie, was, wann und wann nicht?Am Soc Clin Oncol Educ Book.2017: 160-9.
23. Liu H, Meng X, Wang J. Quantitative Echtzeitmethylierung
Nachweis des PAX1-Gens bei der Früherkennung von Gebärmutterhalskrebs.IntJ Gynäkologischer Krebs.2020;30: 1488-92.
24. Imperiale TF, RansohoffDF, Itzkowitz SH, Levin TR, Lavin P, Lidgard GP, et al.Multitargetstool-DNA-Test zur Darmkrebsvorsorge.N EnglJ Med.2014;370: 1287-97.
25. Li J, Li Y, Li W, Luo H, Xi Y, Dong S, et al.Führungspositionierung
Die Sequenzierung identifiziert abweichende DNA-Methylierungsmuster, die die Zellidentität und Tumor-Immunüberwachungsnetzwerke verändern.Genom
Res.2019;29: 270-80.
26. Gao Q, LinYP, Li BS, Wang GQ, Dong LQ, Shen BY, et al.Unaufdringlicher Multikrebsnachweis durch zirkulierende zellfreie DNA-Methylierungssequenzierung (THUNDER): Entwicklung und unabhängige Validierungsstudien.Ann Oncol.2023;34: 486-95.
27. Dong S, Li W, Wang L, Hu J, Song Y, Zhang B, et al.Histonbezogene Gene sind bei Lungenkrebs hypermethyliert und hypermethyliert
HIST1H4F könnte als Pan-Cancer-Biomarker dienen.Krebs Res.2019;79: 6101-12.
28. HeijnsdijkEA, Wever EM, AuvinenA, Hugosson J, Ciatto S, Nelen V, et al.Auswirkungen des Prostata-spezifischen Antigen-Screenings auf die Lebensqualität.N EnglJ Med.2012;367: 595-605.
29. LuzakA, Schnell-Inderst P, Bühn S, Mayer-Zitarosa A, Siebert U. Klinische Wirksamkeit von Krebs-Screening-Biomarkertests, die als selbstzahlende Gesundheitsdienstleistung angeboten werden: eine systematische Überprüfung.Eur J Öffentliche Gesundheit.2016;26: 498-505.
30. Dong S, Lu Q, Xu P, Chen L, Duan X, Mao Z, et al.
Hypermethyliertes PCDHGB7 als universeller Krebsmarker und seine Anwendung bei der Früherkennung von Gebärmutterhalskrebs.Clin Transl Med.2021;11: e457.
31. Yuan J, Mao Z, Lu Q, Xu P, Wang C, Xu X, et al.Hypermethyliertes PCDHGB7 als Biomarker zur Früherkennung von Endometriumkrebs in Endometriumbürstenproben und Gebärmutterhalskrebs.Front Mol Biosci.2022;8: 774215.
32. Dong S, Yang Z, Xu P, Zheng W, Zhang B, Fu F, et al.Gegenseitig
exklusive epigenetische Modifikation von SIX6 mit Hypermethylierung zur Verfolgung des präkanzerösen Stadiums und der Entstehung von Metastasen.Signaltransduktionsziel Ther.2022;7: 208.
33. Huang L, Guo Z, Wang F, Fu L. KRAS-Mutation: von nicht medikamentös zu medikamentös einsetzbar bei Krebs.Signaltransduktionsziel Ther.2021;6: 386.
34. Belinsky SA, Nikula KJ, PalmisanoWA, MichelsR, SaccomannoG, GabrielsonE, et al.Aberrante Methylierung von p16(INK4a) ist ein frühes Ereignis bei Lungenkrebs und ein potenzieller Biomarker für die Früherkennung.Proc Natl Acad Sci U SA.1998;95: 11891-6.
35. Robertson KD.DNA-Methylierung und menschliche Krankheiten.Nat Rev Genet.2005;6: 597-610.
36. WentzensenN, Walker JL, Gold MA, Smith KM, ZunaRE,
Mathews C, et al.Mehrfachbiopsien und Nachweis von Vorstufen von Gebärmutterhalskrebs bei der Kolposkopie.J Clin Oncol.2015;33: 83-9.
37. De Strooper LM, Meijer CJ, Berkhof J, Hesselink AT, Snijders
PJ, Steenbergen RD, et al.Methylierungsanalyse des FAM19A4
Das Gen in Gebärmutterhalskratzern ist äußerst effizient bei der Erkennung von Gebärmutterhalskrebs
Karzinome und fortgeschrittene CIN2/3-Läsionen.Krebs Prev Res (Phila).2014;7: 1251-7.
38. Thai AA, Solomon BJ, Sequist LV, Gainor JF, Heist RS.Lungenkrebs.Lanzette.2021;398: 535-54.
39. Grunnet M, Sorensen JB.Karzinoembryonales Antigen (CEA) als Tumormarker bei Lungenkrebs.Lungenkrebs.2012;76: 138-43.
40. Wood DE, KazerooniEA, Baum SL, EapenGA, EttingerDS, Hou L, et al.Lungenkrebs-Screening, Version 3.2018, NCCN Clinical Practice Guidelines in Oncology.J Natl Compr Canc Netw.2018;16: 412-41.
41. Amerikanische Krebsgesellschaft.Fakten und Zahlen zum Thema Krebs.Atlanta, GA, USA: American Cancer Society;2023 [aktualisiert am 1. März 2023;zitiert am 22. August 2023].
42. FonthamETH, Wolf AMD, Church TR, EtzioniR, Flowers CR,
Herzig A, et al.Gebärmutterhalskrebs-Screening für Personen mit durchschnittlichem Risiko: Richtlinienaktualisierung 2020 der American Cancer Society.CA Cancer J Clin.2020;70: 321-46.
43. Clarke MA, Long BJ, Del Mar MorilloA, Arbyn M, Bakkum-Gamez JN, Wentzensen N. Zusammenhang zwischen Endometriumkrebsrisiko und postmenopausalen Blutungen bei Frauen: eine systematische Überprüfung und Metaanalyse.JAMA Intern Med.2018;178: 1210-22.
44. Jacobs I, Gentry-MaharajA, Burnell M, ManchandaR, Singh N,
Sharma A et al.Empfindlichkeit des transvaginalen Ultraschallscreenings
für Endometriumkarzinom bei postmenopausalen Frauen: eine Fall-Kontroll-Studie innerhalb der UKCTOCS-Kohorte.Lancet Oncol.2011;12: 38-48.
45. BabjukM, Burger M, CompératEM, Gontero P, MostafidAH,
PalouJ, et al.Leitlinien der European Association of Urology zu nicht-muskelinvasivem Blasenkrebs (TaT1 und Carcinoma In Situ) –
Aktualisierung 2019.Eur Urol.2019;76: 639-57.
46. Aragon-Ching JB.Herausforderungen und Fortschritte in der Diagnose, Biologie und Behandlung von Karzinomen des oberen Urotheltrakts und der Blase.Urol Oncol.2017;35: 462-4.
47. Rizvi S, KhanSA, Hallemeier CL, Kelley RK, Gores GJ.
Cholangiokarzinom – sich entwickelnde Konzepte und Therapiestrategien.Nat Rev Clin Oncol.2018;15: 95-111.
48. Ye Q, Ling S, Zheng S, Xu X. Flüssigbiopsie in hepatozellulären
Karzinom: zirkulierende Tumorzellen und zirkulierende Tumor-DNA.Mol-Krebs.2019;18: 114.
49. Zhang Y, Yao Y, Xu Y, Li L, Gong Y, Zhang K, et al.Pan-Krebs
Nachweis zirkulierender Tumor-DNA bei über 10.000 chinesischen Patienten.Nat Commun.2021;12: 11.
50. Eisenhauer EA, Therasse P, BogaertsJ, Schwartz LH, Sargent D, Ford R, et al.Neue Kriterien zur Bewertung des Ansprechens bei soliden Tumoren: überarbeitete RECIST-Leitlinie (Version 1.1).Eur J Krebs.2009;45: 228-47.
51. LitièreS, Collette S, de Vries EG, Seymour L, BogaertsJ.RECIST – Aus der Vergangenheit lernen, um die Zukunft zu gestalten.Nat Rev Clin Oncol.
2017;14: 187-92.
52. Seymour L, BogaertsJ, Perrone A, FordR, Schwartz LH, Mandrekar S, et al.iRECIST: Richtlinien für Antwortkriterien zur Verwendung in Studien
Testen von Immuntherapeutika.Lancet Oncol.2017;18: e143-52.
53. PantelK, Alix-Panabières C. Flüssigbiopsie und minimale Resterkrankung – neueste Fortschritte und Auswirkungen auf die Heilung.Nat Rev Clin Oncol.2019;16: 409-24.
Zitieren Sie diesen Artikel als: Qian C, Zou X, Li W, Li Y, Yu W. Der Vorposten gegen Krebs: Universelle Krebsmarker.Cancer Biol Med.2023;20: 806-815.
doi: 10.20892/j.issn.2095-3941.2023.0313
Zeitpunkt der Veröffentlichung: 07.05.2024